Principe de l'électrophorèse sur gel en gradient dénaturant (DGGE)
L'électrophorèse sur gel en gradient dénaturant (DGGE) est une technique de prise d'empreinte indépendante de la culture qui permet une analyse comparative rapide des modifications des communautés microbiennes. Les gènes d’ARNr 16S amplifiés à partir d’échantillons de l’environnement peuvent être séparés en fonction de leur comportement de fusion dans un gradient dénaturant de l’urée et du formamide. Une empreinte digitale de la communauté microbienne est générée, chaque bande sur le gel étant supposée correspondre à une espèce bactérienne différente. La dynamique communautaire peut ensuite être évaluée par l'analyse statistique des profils DGGE et le séquençage des bandes excisées.
Les communautés microbiennes sont des systèmes hautement dynamiques pouvant présenter des variations spatiales et temporelles et capables de réagir au stress environnemental. Comprendre la dynamique d'une communauté est une étape importante pour pouvoir faire des prévisions précises concernant le comportement de la communauté ou pour obtenir des résultats particuliers. Cependant, la capacité à surveiller les changements dans la composition des communautés microbiennes nécessite une technique permettant de comparer rapidement une série d’échantillons. Bien que des approches telles que le séquençage de gènes clonés fournissent des informations phylogénétiques détaillées, elles ne sont pas pratiques pour comparer simultanément un grand nombre d'échantillons.
Les techniques d’empreintes digitales communautaires, y compris l’électrophorèse sur gel en gradient dénaturant (DGGE), reposent sur l’analyse rapide de gènes marqueurs récupérés par PCR (souvent des ARNr 16S) et se sont révélées être des outils précieux pour générer rapidement des profils de diversité afin de caractériser un éventail de communautés microbiennes.
Bien que cette technique soit considérée comme permettant de détecter des micro-organismes dont l'abondance est supérieure à 1%, les populations moins abondantes peuvent être analysées à l'aide d'analyses DGGE spécifiques à un groupe, soit par une approche PCR imbriquée, soit à l'aide d'amorces spécifiques à un groupe compatibles avec DGGE.
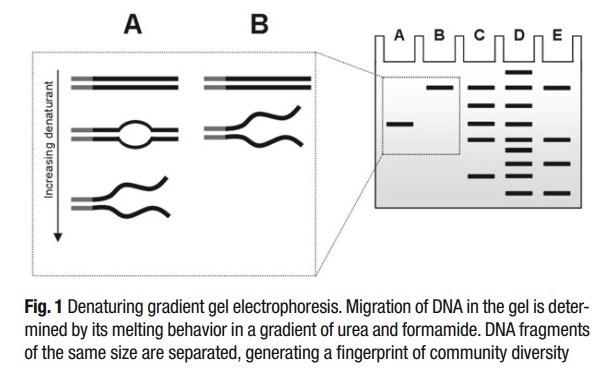
DGGE implique la séparation de fragments d'ADN double brin de même taille mais de séquence différente (Fig. 1). La mobilité des fragments dépend de leur comportement à la fusion dans un gradient dénaturant d'urée et de formamide. Au fur et à mesure que les fragments migrent le long du gel de Polyacrylamide, ils rencontrent des concentrations de plus en plus importantes de dénaturants. Les différences de séquence entraînent la fusion des fragments à différentes positions le long du gradient. Les fragments partiellement dénaturés vont migrer plus lentement que les fragments non dénaturés. Afin d'empêcher les deux brins d'ADN de se dissocier complètement et de détecter les mutations dans les domaines de fusion plus élevée, un fragment riche en GC (pince GC) est ajouté à l'une des amorces de PCR. En conséquence, différentes séquences migreront à différentes distances le long du gel, générant un profil DGGE caractéristique de la communauté microbienne. Lors de l'interprétation des profils DGGE, il est supposé que chaque bande correspond à une espèce bactérienne différente. Toutefois, il convient de garder à l’esprit que certaines bandes peuvent être composées de plus d’une espèce co-migratrice ou que certaines espèces peuvent contribuer à plus d’une bande si elles possèdent plusieurs copies différentes du gène de l’ARNr 16S.

Plusieurs profils DGGE peuvent être comparés pour observer des différences spatiales ou temporelles au sein d'une communauté microbienne (Fig. 2). Cette technique a déjà été utilisée pour étudier les changements temporels survenant dans le microbiote intestinal de mammifères ainsi que dans les communautés du sol. L'un des principaux avantages du DGGE, comparé à d'autres techniques d'empreinte digitale telles que le polymorphisme de longueur des fragments de restriction terminaux (T-RFLP), est que l'identité des bandes dans le gel peut être déterminée par excision et séquençage. Par conséquent, il est possible de déterminer l'identité des bactéries impliquées dans les modifications de la composition de la communauté.
Protocole de la technique DGGE
- Matériels et méthodes de DGGE
- Extraction d'ADN plasmidique bactérien
- Purification et Contrôle de la qualité de l'ADN
- Extraction d'ADN à partir de racines et des feuilles de plantes
- Extraction d'ADN de sols, de sédiments, de biofilms et de champignons
- Comment choisir les séquences d'amorce d'ADN ?
- Processus d'amplification de l'ADN par PCR
- Extraction d'ADN plasmidique bactérien
- Extraction d'ADN plasmidique par lyse alcaline rôle des réactifs
