Réaction de Polymérase en Chaine (PCR)
La PCR est une technique utilisée pour produire de grandes quantités d' ADN dans une région définie, qui est flanquée de deux amorces.
On parle d'amplification des acides nucléiques. le mot PCR est une abbréviation de (Polymerase Chain Reaction) Amplification en chaîne par polymérase (PCR), cette technique est développée par Kary Mullis en 1985.
Qu’est-ce une amorce d’ADN ?
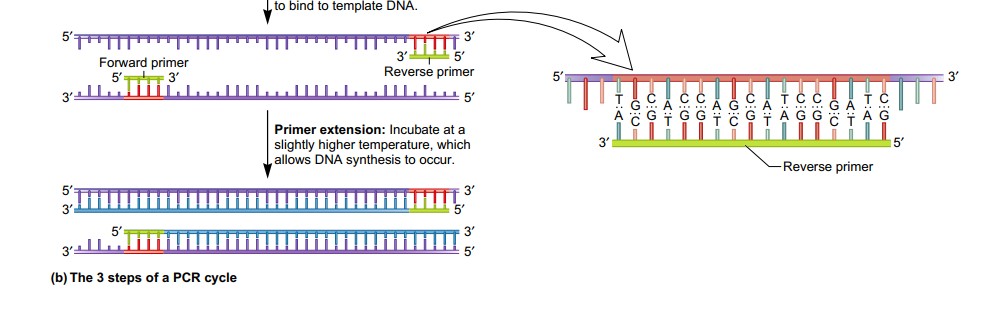
Les amorces sont des oligonucléotides, qui sont de courts segments d'ADN, généralement d'environ 15 à 20 nucléotides de long.
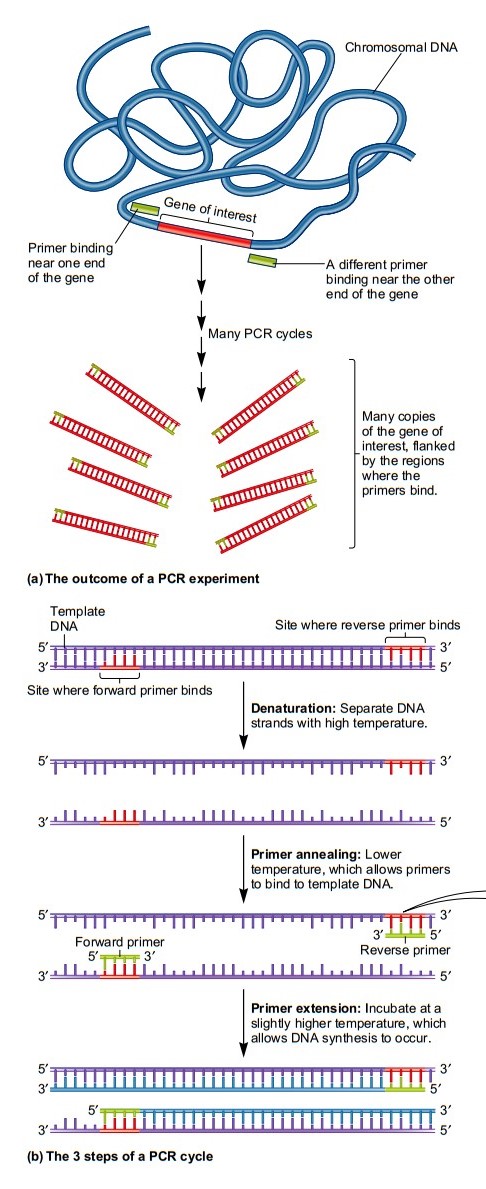
Processus d’amplification d’ADN
Comme le montre la figure, le matériau de départ d'une expérience de PCR peut être un mélange complexe d'ADN. Les deux amorces se lient à des sites spécifiques de l'ADN car leurs bases sont complémentaires sur ces sites. Le résultat final de la PCR est que la région qui est flanquée par les amorces, qui contient le gène d'intérêt, est amplifiée. Le terme amplification signifie que chaque cycle de PCR se déroule en trois étapes: dénaturation, hybridation d'amorce et extension ou élongation d'amorce. Les amorces utilisées dans les expériences de PCR réelles ont généralement une longueur d'environ 15 à 20 nucléotides. La région entre les deux amorces a généralement une longueur de centaines de nucléotides.
De nombreux exemplaires de la région ont été réalisés. En d'autres termes, la région entre les deux amorces a été clonée. Les amorces sont des réactifs clés dans une expérience de PCR. Les amorces sont fabriquées chimiquement. Ils ne sont généralement pas fabriqués dans des laboratoires de recherche ou des laboratoires cliniques, mais dans des établissements universitaires ou industriels. Les chercheurs ou les cliniciens commandent simplement les amorces dont ils ont besoin avec une séquence spécifiée.
Le choix des amorces pour PCR
Dans la plupart des cas, la PCR est effectuée sur des échantillons d'ADN dans lesquels un scientifique connaît déjà les séquences d'ADN qui bordent la région d'intérêt. Comme le génome entier de nombreuses espèces a déjà été déterminé, les informations sur les séquences peuvent provenir de la base de données génomique des espèces d'intérêt. Par exemple, si vous souhaitez cloner un gène humain ou une partie du gène humain, tel que le gène de la β globine, vous rechercheriez cette séquence génétique dans la base de données du génome humain et décideriez de la liaison de vos deux amorces. Vous pouvez alors commander des amorces avec des séquences complémentaires à ces sites.
Quel sont les réactifs de PCR ?
Considérons maintenant les réactifs nécessaires pour une expérience de PCR. Les deux réactifs que nous avons déjà examinés à la figure 18.5a sont les amorces et un échantillon d'ADN contenant un gène d'intérêt, appelé matrice ADN. De plus, des désoxyribonucléosides triphosphates (dNTP) sont nécessaires, de même qu'une forme thermostable de l'ADN polymérase telle que la Taq polymérase, isolée de la bactérie Thermus aquaticus. Une forme thermostable de l'ADN polymérase est nécessaire car la PCR implique des étapes de chauffage qui inactivent la plupart des autres formes naturelles d'ADN polymérase (qui sont thermolabiles ou facilement dénaturées par la chaleur).
Les étapes de la PCR
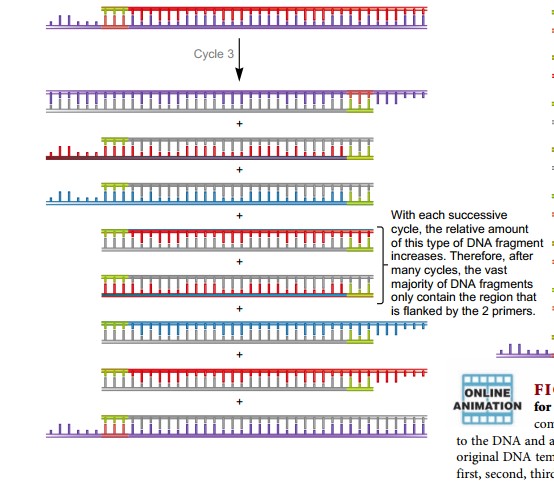
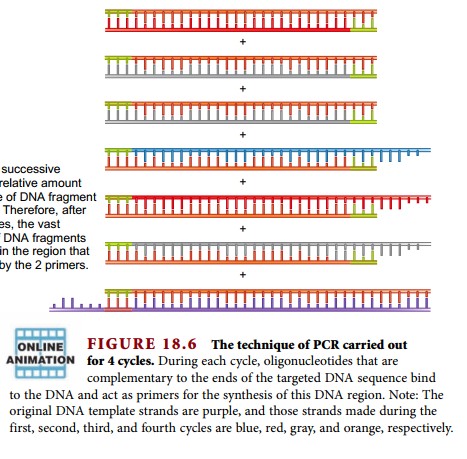
La technique de PCR implique trois (3) étapes indispensable, On parle de Dénaturation, hybridation de l'amorce et extension ou élongation de l'amorce. Pour faire des copies de l'ADN, l'ADN matrice est d'abord dénaturé par traitement thermique, provoquant la séparation des brins. Lorsque la température est abaissée, les amorces oligonucléotidiques se lient à l'ADN dans un processus appelé recuit. Une fois que les amorces ont été recuites, la température est légèrement élevée et la Taq polymérase catalyse la synthèse de brins d'ADN complémentaires dans la direction 5ʹ à 3ʹ, en commençant par les amorces. Ce processus, appelé extension de l'amorce, double la quantité d'ADN de la matrice. Les trois étapes représentées sur la figure 18.5b constituent un cycle d'une réaction de PCR. Le processus séquentiel de dénaturation - recuit d'amorce - extension d'amorce est ensuite répété pendant plusieurs cycles pour doubler la quantité d'ADN matrice plusieurs fois de suite.
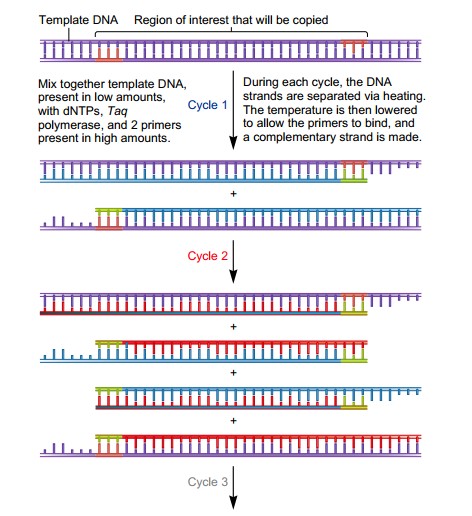
Cette méthode est appelée une réaction en chaîne car les produits de chaque réaction précédente (les brins d'ADN nouvellement fabriqués) sont utilisés comme réactifs (les brins de matrice) dans les réactions ultérieures. La figure 18.6 suit une expérience de PCR sur quatre cycles. Comme la région d'intérêt est doublée à chaque cycle, le résultat final de quatre cycles correspond à 24 ou 16 copies de la région d'intérêt.

Appareillage
Étant donné que le matériau de départ contient de longs brins d'ADN chromosomique, certains des produits d'une expérience de PCR contiennent la région d'intérêt, ainsi que de l'ADN supplémentaire à chaque extrémité. Cependant, après de nombreux cycles de PCR, les produits ne contenant que la région d'intérêt prédominent fortement dans le mélange.
La PCR est effectuée dans un réacteur thermique, appelé thermocycleur, qui automatise la synchronisation de chaque cycle.

L'expérimentateur mélange l'échantillon d'ADN, les dNTP, la Taq polymérase et un excès d'amorces ensemble dans un seul tube dite (mixte de PCR). Le tube est placé dans un thermocycleur et l'expérimentateur configure la machine pour qu'elle fonctionne dans une plage de températures et un nombre de cycles définis.
Au cours de chaque cycle, le thermocycleur augmente la température pour dénaturer les brins d'ADN, puis abaisse la température pour permettre le recuit et l'extension. Typiquement, chaque cycle dure 2 à 3 minutes et est ensuite répété. Une analyse PCR typique impliquera probablement 20 à 30 cycles de réplication.

0 Commentaires