Principe d'extraction d'ADN des bactéries à Gram positive
Les bactéries à Gram positif ont une paroi rigide avec une épaisse couche de pepidoglycane qui rend la lyse de la cellule difficile.
Les bactéries lactiques, les staphylocoques ou les actinomycètes sont des bactéries à Gram positif. L'extraction de l'ADN par des solvants n'est donc pas toujours rentable et la quantité d'ADN est faible.
Dans cet article, nous nous concentrons sur une technique hybride (utilisant la TE concentrée avec ébullition) et rapide pour l'extraction de l'ADN des bactéries à gram positif.
L'extraction d' ADN consiste à libérer le matériel génétique de la cellule dans le milieu. Il existe plusieurs techniques et méthodes de lyse cellulaire qui aboutit à l'extraction de l'adn génomique d'une cellule.
L'objectif principal de cette expérience est de fournir un ADN de haute qualité pouvant être stocké pendant plusieurs années dans des conditions appropriées.
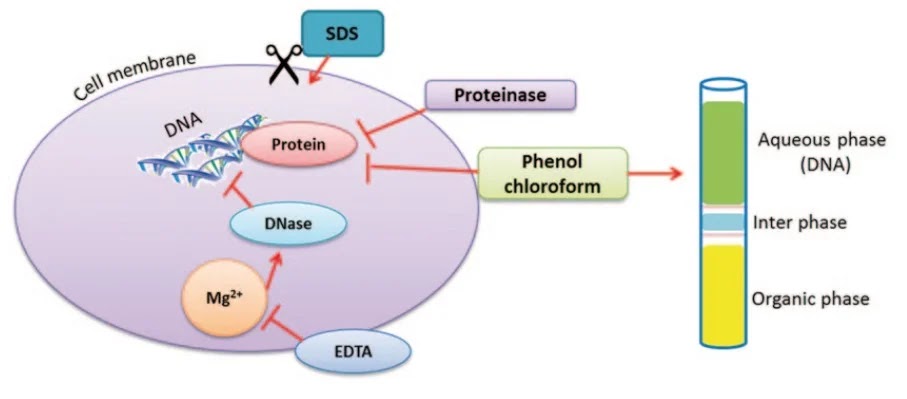
Principe de Protocole d'extraction d'ADN
Dans le protocole suivant, une technique très simple, rentable et rapide est utilisée. Tout d'abord, nous utilisons l'ébullition dans un tampon TE plus ou moins concentré (tris-edta pH=8) pour dégrader les parois cellulaires, puis nous utilisons de l'isopropanol et de l'éthanol à 70 % pour la purification. Après élimination de l'éthanol, l'ADN de haute qualité est remis en suspension dans un tampon à faible TE. Le rapport A260/A280 est toujours de 1,8-2 grâce à l'EDTA et la quantité finale d'ADN varie entre 40 ng/µl et 500 ng/µl.
Proctocole d'extaction d'ADN bactérien
- Inoculer une seule colonie bactérienne dans 5 ml de milieu de bouillon LB et incuber les tubes à 37 ° C pendant 24 h sous agitation à 180 tr / min.
- **La periode d'incubation dépend du type de la bactérie
- Recueillir le culot cellulaire bactérien par centrifugation à 6000 tr / min pendant 5 min à température ambiante.
- Transférer le culot dans des tubes Eppendorf 1.5 ml
- Jeter le surnageant et remettre en suspension le culot cellulaire dans 250 µl du High TE.
- Ajouter 250 µl du High TE (Tris-EDTA, pH=8) (20mM Tris, 10 mM EDTA)
- Il faut bien mélanger la suspension
- Régler le bain-marie à 100 °C ou faites bouillir de l'eau dans un récipient.
- Gardez les tubes à centrifuger contenant la suspension bactérienne dans de l'eau bouillante pendant 10 min.
- Enclenchez immédiatement les tubes à centrifuger dans de la glace pendant 5 min.
- Après incubation dans de la glace, centrifuger le tube à 10000 tr/min pendant 5 min à 4 °C.
- Transférer le surnageant (120 µl) dans un nouveau tube.
- Ajouter 2/3 le volume de l'isopropanol, (80µl)
- Mélanger par inversement tout doucement 8-10 fois
- Vous pouvez mettre le tube dans du la glace à 30 min
- Centrifuger à 10 000 tr / min pendant 10 min dans la Température ambiante.
- Eliminer le surnageant et ajouter 100 à 150 µl d'éthanol à 70% pour obtenir un culot.
- Faire pippter par va-et-vien pour laver l'ADN.
- Répéter l'étape de lavaage avec du l'etanol 70% si necessaire
- Centrifuger ce mélange pendant 5 min à la température ambiante, et sécher le culot par évaporation complète de l'éthanol.
- Ajouter de Low TE (10 mM Tris, 1mM EDTA, pH=8)
- Doser l'ADN par Nanodrop ou par gel d'electrophorèse

0 Commentaires